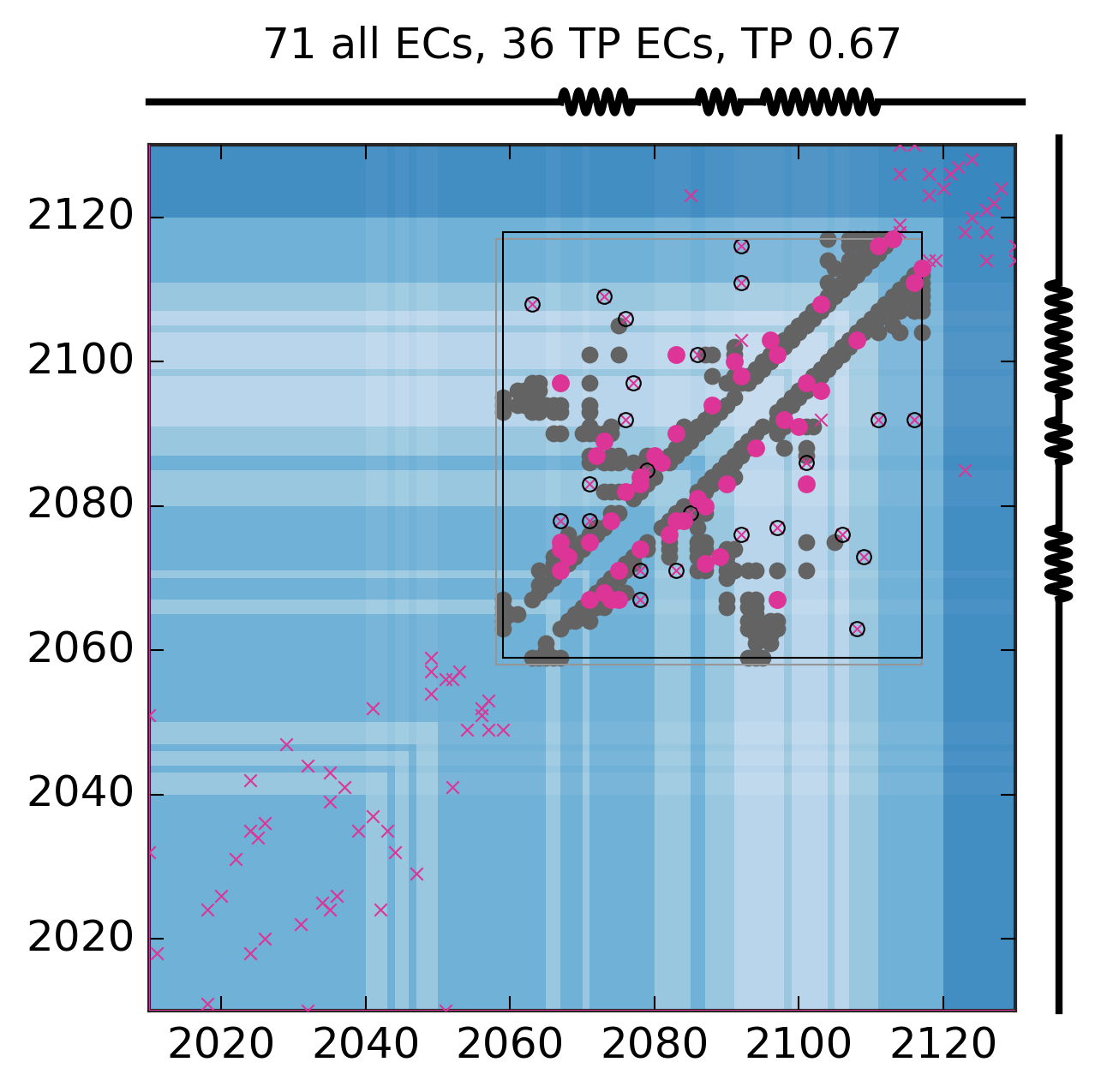
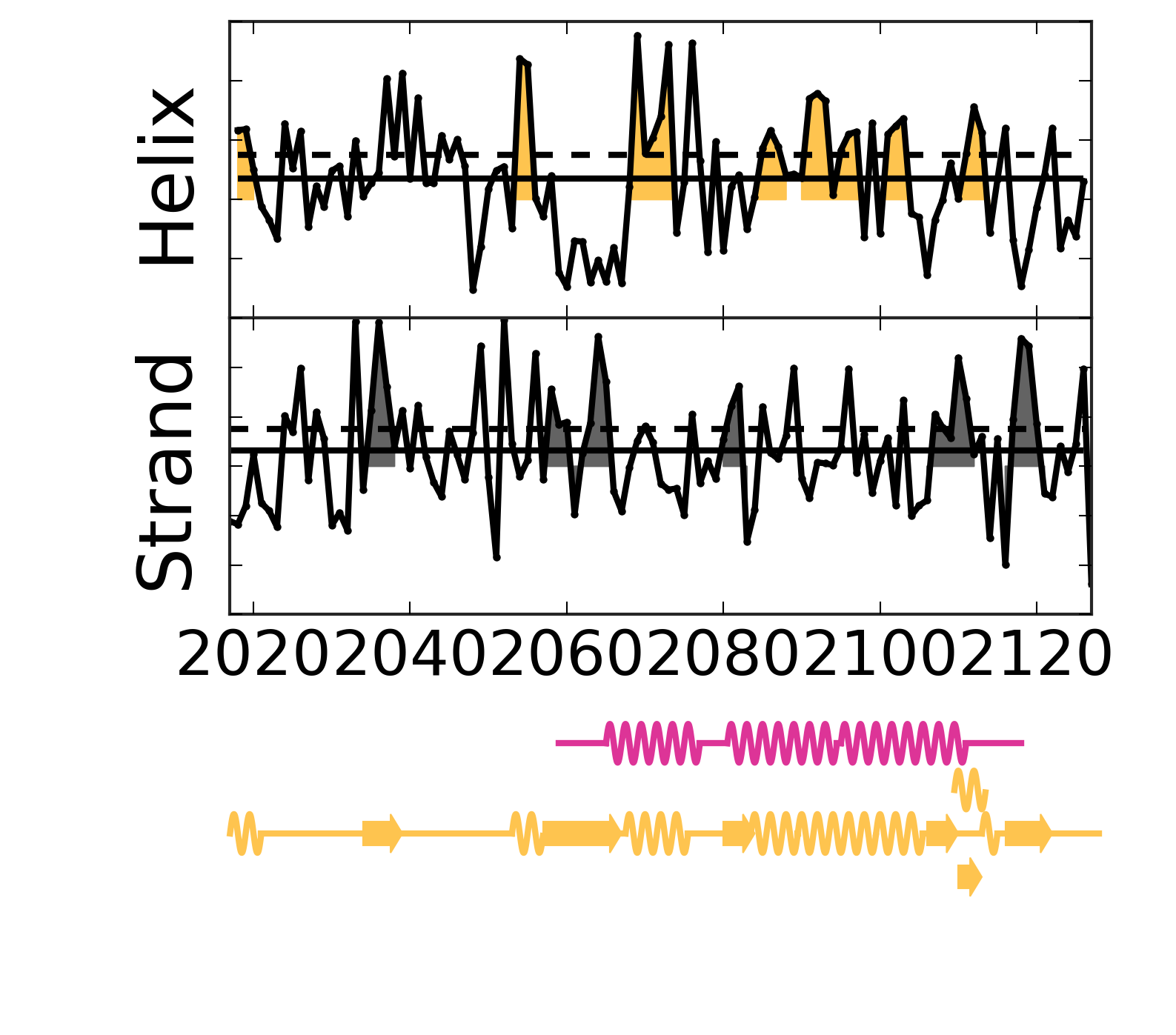
Evolutionary Coupling Analysis
Predicted and experimental contacts
Secondary structure from ECs
Known pdb structures
| pdb |
chain |
| 1jjs |
A |
| 1kbh |
B |
| 2c52 |
A |
| 2kkj |
A |
| 2l14 |
A |
EC score distribution and threshold
Top ECs
| Rank |
Residue 1 |
Amino acid 1 |
Residue 2 |
Amino acid 2 |
EC score |
| 1 |
2121 |
G |
2126 |
P |
0.22 |
| 2 |
2077 |
S |
2097 |
L |
0.20 |
| 3 |
2049 |
V |
2054 |
M |
0.20 |
| 4 |
2096 |
Q |
2103 |
K |
0.20 |
| 5 |
2076 |
K |
2092 |
K |
0.18 |
| 6 |
2114 |
P |
2118 |
P |
0.17 |
| 7 |
2067 |
A |
2071 |
L |
0.17 |
| 8 |
2120 |
P |
2124 |
S |
0.16 |
| 9 |
2071 |
L |
2078 |
P |
0.16 |
| 10 |
2092 |
K |
2111 |
A |
0.16 |
| 11 |
2049 |
V |
2059 |
P |
0.15 |
| 12 |
2081 |
P |
2086 |
Q |
0.15 |
| 13 |
2051 |
G |
2056 |
N |
0.15 |
| 14 |
2076 |
K |
2106 |
T |
0.15 |
| 15 |
2053 |
R |
2057 |
V |
0.15 |
| 16 |
2037 |
P |
2041 |
M |
0.15 |
| 17 |
2025 |
A |
2034 |
P |
0.15 |
| 18 |
2114 |
P |
2130 |
P |
0.15 |
| 19 |
2103 |
K |
2108 |
K |
0.14 |
| 20 |
2113 |
Q |
2117 |
Q |
0.14 |
| 21 |
2049 |
V |
2057 |
V |
0.14 |
| 22 |
2086 |
Q |
2101 |
F |
0.14 |
| 23 |
2010 |
S |
2051 |
G |
0.14 |
| 24 |
2080 |
S |
2087 |
V |
0.13 |
| 25 |
2078 |
P |
2083 |
Q |
0.13 |
| 26 |
2118 |
P |
2126 |
P |
0.13 |
| 27 |
2020 |
G |
2026 |
P |
0.13 |
| 28 |
2041 |
M |
2052 |
P |
0.13 |
| 29 |
2122 |
L |
2127 |
G |
0.13 |
| 30 |
2032 |
P |
2044 |
Q |
0.13 |
| 31 |
2114 |
P |
2119 |
Q |
0.13 |
| 32 |
2072 |
L |
2087 |
V |
0.13 |
| 33 |
2074 |
T |
2078 |
P |
0.13 |
| 34 |
2067 |
A |
2075 |
L |
0.13 |
| 35 |
2071 |
L |
2075 |
L |
0.13 |
| 36 |
2079 |
S |
2085 |
Q |
0.13 |
| 37 |
2092 |
K |
2098 |
M |
0.13 |
| 38 |
2029 |
Q |
2047 |
A |
0.13 |
| 39 |
2024 |
Q |
2042 |
S |
0.12 |
| 40 |
2078 |
P |
2084 |
Q |
0.12 |
| 41 |
2022 |
W |
2031 |
Q |
0.12 |
| 42 |
2026 |
P |
2036 |
M |
0.12 |
| 43 |
2091 |
L |
2100 |
A |
0.12 |
| 44 |
2085 |
Q |
2123 |
Q |
0.12 |
| 45 |
2083 |
Q |
2090 |
I |
0.12 |
| 46 |
2097 |
L |
2101 |
F |
0.12 |
| 47 |
2010 |
S |
2032 |
P |
0.12 |
| 48 |
2035 |
G |
2039 |
P |
0.12 |
| 49 |
2083 |
Q |
2101 |
F |
0.12 |
| 50 |
2067 |
A |
2074 |
T |
0.12 |
| 51 |
2071 |
L |
2083 |
Q |
0.12 |
| 52 |
2024 |
Q |
2035 |
G |
0.12 |
| 53 |
2011 |
G |
2018 |
P |
0.12 |
| 54 |
2088 |
L |
2094 |
N |
0.12 |
| 55 |
2067 |
A |
2078 |
P |
0.12 |
| 56 |
2052 |
P |
2056 |
N |
0.12 |
| 57 |
2118 |
P |
2123 |
Q |
0.12 |
| 58 |
2114 |
P |
2126 |
P |
0.12 |
| 59 |
2018 |
P |
2024 |
Q |
0.12 |
| 60 |
2035 |
G |
2043 |
M |
0.12 |
| 61 |
2063 |
I |
2108 |
K |
0.12 |
| 62 |
2124 |
S |
2128 |
M |
0.12 |
| 63 |
2068 |
L |
2073 |
R |
0.12 |
| 64 |
2116 |
M |
2130 |
P |
0.11 |
| 65 |
2092 |
K |
2116 |
M |
0.11 |
| 66 |
2073 |
R |
2089 |
N |
0.11 |
| 67 |
2067 |
A |
2097 |
L |
0.11 |
| 68 |
2076 |
K |
2082 |
Q |
0.11 |
| 69 |
2111 |
A |
2116 |
M |
0.11 |
| 70 |
2073 |
R |
2109 |
Y |
0.11 |
| 71 |
2092 |
K |
2103 |
K |
0.11 |
Alignment robustness analysis
First most common residue correlation
Second most common residue correlation